巴氏吸管由醫用級聚乙烯(PE)制成,管體
施一公組報道釀酒酵母pre-B complex狀態剪接體電鏡結構
| 導讀 | 2018年5月25日,清華大學生命科學學院、結構生物學高精尖創新中心施一公教授研究組就剪接體的組裝機理與結構研究于《科學》(Science)雜志以長文形式再次發表重大研究成果。 |

本文轉載自“結構生物學高精尖創新中心”,原標題:施一公研究組在《科學》發文報道處于激活前狀態的兩個*組裝的釀酒酵母剪接體高分辨率電鏡結構。
這篇題為《*組裝的釀酒酵母剪接體激活前結構》(Structures of the Fully Assembled Saccharomyces cerevisiae Spliceosome Before Activation)的論文報道了釀酒酵母剪接體處于被激活前階段的兩個*組裝的關鍵構象——預催化剪接體前體(precursor pre-catalytic spliceosome,定義為“pre-B復合物”)和預催化剪接體(pre-catalytic spliceosome,定義為“B復合物”)。這兩個整體分辨率分別為3.3-4.6埃和3.9埃的高分辨率三維結構展示了在剪接體組裝過程中pre-mRNA的5’剪接位點和分支點(BPS)的識別狀態與動態變化,回答了剪接體激活前pre-mRNA的5’剪接位點和分支點識別機理,以及激活過程中5’剪接位點和分支點如何逐步進入活性位點、剪接體如何逐步組裝并通過結構重組zui終完成激活等重要問題。
RNA剪接是真核生物基因表達調控的重要環節之一。上世紀70年代,科學家們發現真核生物基因的不連續性,從而表明遺傳信息從DNA轉移到RNA上之后,需要經歷有效遺傳信息的“剪斷”與重新“拼接”,這種有效遺傳信息的拼接與“無效”遺傳信息的去除,被稱為RNA剪接。RNA剪接普遍存在于真核生物中,隨著物種的進化,含有內含子的基因數量增加,發生RNA剪接的頻率也相應增高,使得一個基因編碼多個蛋白質成為可能。RNA剪接的本質是兩步轉酯反應,這種看似簡單的化學反應在細胞中難以自行發生,而負責執行這一化學反應的是細胞核內一個巨大且高度動態變化的分子機器——剪接體(spliceosome)。在剪接反應過程中,多種蛋白質-核酸復合物及剪接因子按照高度的順序發生結合和解聚,依次形成預組裝復合物U4/U6.U5 Tri-snRNP(U4/U6.U5三小核核糖核蛋白復合物)以及至少8個狀態的剪接體pre-B、B、Bact、B*、C、C*、P以及ILS復合物。
由于剪接體高度的動態性和復雜性,獲得不同狀態的剪接體的高分辨率三維結構被*為世界難題。在這種巨大的挑戰下,施一公教授率領研究組迎難而上,經過7年的努力,終于在2015年報道了裂殖酵母剪接體3.6埃的高分辨率結構,展示了剪接體催化中心近原子分辨率的結構。這一重大研究成果對RNA剪接機理的研究產生革命性影響。自2015年*個剪接體結構發表以后,施一公研究組相繼解析了釀酒酵母剪接體復合物處于6個不同狀態的高分辨率結構,分別是3.8埃的預組裝復合物U4/U6.U5 Tri-snRNP、3.5埃的激活狀態復合物Bact complex、3.4埃的*步催化反應后復合物C complex、4.0埃的第二步催化激活狀態下的C* complex、3.6埃的完成兩步轉酯反應后狀態下的P complex,以及3.5埃的內含子套索剪接體ILS complex的結構。這些已解析的剪接體基本覆蓋了整個RNA剪接循環,從分子層面揭示了剪接體催化RNA剪接兩步反應的工作機理,同時為理解剪接體的激活和解聚等過程的發生提供依據。然而,想要清楚解釋剪接體是如何逐步組裝并完成激活的機制仍有困難,而新發表的這篇文章所解析的兩個關鍵狀態的剪接體,則彌補了領域內對這一部分研究的缺陷。
本文報道的處于激活前的兩個*組裝的剪接體結構,從復合物的提純、樣品的制備到結構的解析,每一步都十分具有挑戰。預催化剪接體前體(pre-B complex)由U1 snRNP、U2 snRNP以及U4/U6.U5 tri-snRNP組成,目前被認為是組成蛋白zui多、分子量zui大的剪接體,該狀態結構復雜,但各組分之間的相互作用并不緊密,使得該復合物在提純過程中十分容易解聚。在新發表的這篇《科學》文章中,施一公研究組對提純方案多次探索,zui終優化出一套可以獲得穩定的、性質良好的pre-B complex樣品。隨后利用單顆粒冷凍電鏡技術重構出了U1 snRNP、U2 snRNP以及U4/U6.U5 tri-snRNP部分分辨率高達3.3埃、3.6-4.6埃以及3.4埃的冷凍電鏡結構,并搭建了原子模型(圖1)。
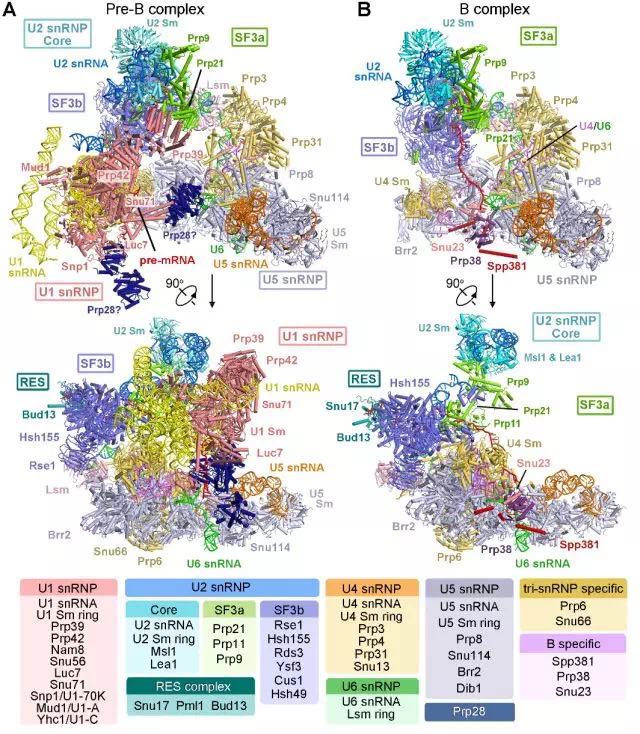
圖1 釀酒酵母預催化剪接體前體和
預催化剪接體的三維結構
該文解析的pre-B complex結構是目前世界上已解析的wei一一個同時包含五種核糖核蛋白(snRNP)剪接體結構,它由68個蛋白和6條RNA組成。在該結構中,觀察到了剪接體組裝早期U1 snRNP對5’剪接位點的識別,以及五種核糖核蛋白之間的相互作用界面。與此同時,該文還報道了處于pre-B complex之后的另一個*組裝的剪接體,即預催化剪接體B complex的高分辨率三維結構。結合B complex的結構信息,通過結構對比,可以清楚的看到在組裝過程中,pre-mRNA的5’剪接位點由一開始被U1 snRNP識別,而后由于構象變化被轉移并與U6 snRNA配對,這一步的變化為剪接體激活提供了結構基礎。除此之外,分支點的動態變化、剪接體的各組分所經歷的結構重組與構象改變也都清晰的呈現出來。在文章zui后,根據pre-B的結構特征,作者還大膽推測了zui早期的不*組裝的預剪接體(pre-spliceosome,定義為“A 復合物”)的三維結構模型(圖2)。這兩個關鍵狀態剪接體結構的解析,為揭示剪接體組裝初期如何識別5’剪接位點和分支點、如何進行結構重組以及如何完成剪接體的激活等問題的機理提供了zui直接、有效的結構證據,也將為更高等真核生物可變剪接的研究提供結構基礎與理論依據。
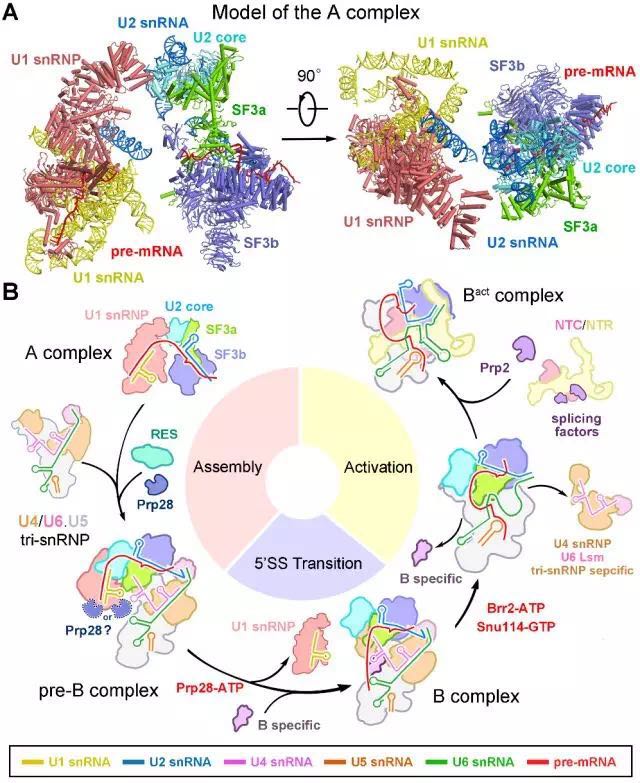
圖2 釀酒酵母預剪接體三維結構的預測與
剪接體組裝并激活的模型
截至目前為止,施一公研究組在酵母中一共解析了9個不同狀態的剪接體高分辨的三維結構(圖3),從組裝到被激活,從發生兩步轉酯反應到剪接體的解聚,這9個狀態的剪接體完整覆蓋了剪接通路,將剪接體介導RNA剪接的過程串聯起來,為理解RNA剪接的分子機理提供了zui清晰、zui全面的結構信息。
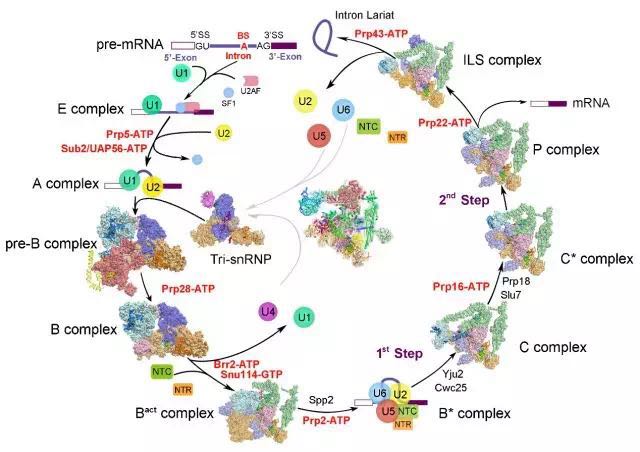
圖3 施一公研究組解析的酵母剪接體結構匯總
(圖片來源: Shi Lab)
清華大學生命科學學院、結構生物學高精尖創新中心施一公教授為本文的通訊作者;清華大學生命科學學院三年級博士研究生白蕊,醫學院博士后、結構生物學高精尖創新中心學者萬蕊雪以及生命科學學院博士后、結構生物學高精尖創新中心學者閆創業為該文的共同*作者;清華大學冷凍電鏡平臺的雷建林博士為冷凍電鏡數據收集提供了幫助。電鏡數據采集于清華大學冷凍電鏡平臺,計算工作得到清華大學高性能計算平臺、國家蛋白質設施實驗技術中心(北京)的支持。本工作獲得了北京市結構生物學高精尖創新中心及國家自然科學基金委的經費支持。
 您好, 歡迎來到化工儀器網
您好, 歡迎來到化工儀器網

 12
12


