聯系電話
- 聯系人:
- 曹女士
- 電話:
- 400-6111-883
- 手機:
- 售后:
- 4006-111-883
- 傳真:
- 86-21-34615995
- 地址:
- 上海市浦東新區天雄路166弄1號3樓
- 網址:
- www.yeasen.com
掃一掃訪問手機商鋪
遺傳中心法則表明,DNA是生物體內遺傳信息的載體。遺傳信息在精密的調控下從DNA轉錄成RNA,再傳遞到蛋白質。因此RNA被認為是DNA與蛋白質之間生物信息傳遞的“橋梁”,在研究轉錄組信息中占有著重大的作用。
背景介紹
轉錄組(transcriptome)是指在某一特定的生理條件下,細胞、組織或者生物體內所有的轉錄產物的集合,即轉錄出來的所有RNA總和,包括編碼RNA(即mRNA)和ncRNA(tRNA、rRNA、miRNA、lncRNA、circRNA)等不同類型的RNA分子。在這之中核糖體RNA (rRNA) 約占RNA總量的 80%,是多的一類RNA,通常這類RNA分子量比較大且代謝不活躍,種類包括:原核生物中5S rRNAs、16S rRNAs和23S rRNAs三種,真核生物中5S rRNAs、5.8S rRNAs、18S rRNAs和28S rRNAs四種。
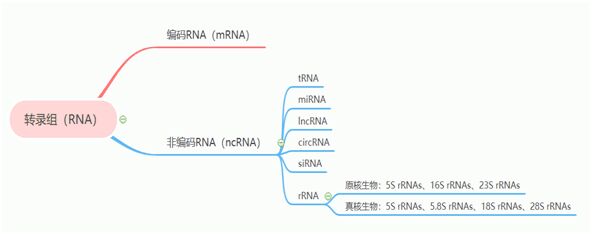
圖1:RNA分類圖
rRNA的“作用”
隨著人類基因組計劃(HGP)和DNA元件百科全書計劃(ENCODE)的完成,人們發現在人類基因組中大部分DNA都可以轉錄成RNA,但是僅有1.5%的核苷酸序列用于蛋白質的編碼,剩余不編碼蛋白質的的非編碼RNA(ncRNA),被認為是基因組轉錄噪音。這種轉錄組噪音(無效信息)基本全部來源于豐度zui高的成員——rRNA。
測序的目的就是為了更多的獲得生物信息,但是rRNA這個在RNA中zui豐富的成員卻只能提供非常少的轉錄本的信息,且檢測到過多的rRNA會掩蓋其它基因的表達豐富度;因此,通常在測序之前從RNA樣品中除去rRNA,rRNA去除的效率也被視為zui大化讀取到轉錄物的關鍵因素。
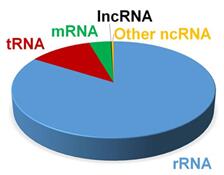
圖2:人類組織或細胞總RNA中各種RNA分布餅狀圖
rRNA的“去除法”
目前rRNA去除的方法主要有兩種,一種是通過DNA探針或者RNA探針雜交捕獲rRNA再用磁珠吸附去除的方法,這種方法稱為Ribo-Zero-seq;另一種是利用雙鏈特異性核酸酶處理的方法提取mRNA,稱為DSN-seq。兩種方法對應的原理與區分如下所示:
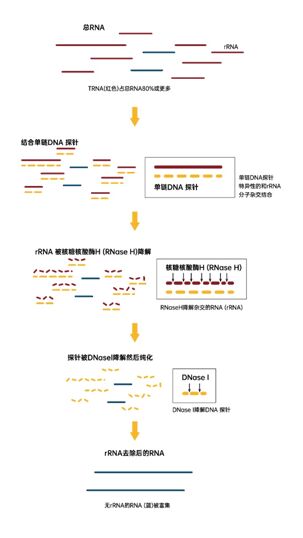
方法一(DSN-seq)
去除流程:特異性探針(區分物種)與rRNAs雜交——RNase H消化rRNAs——DNase I消化探針——其他RNA富集純化
占比:在市場現有品牌中占據絕大多數
優勢:樣本起始量要求低;rRNA殘留量更低
缺點:其操作較復雜;暫無混合樣本效果驗證;
流程圖:
方法二(Ribo-Zero-seq)
去除流程:生物素標記探針與rRNAs雜交——鏈霉親和素磁珠去除探針與rRNAs——其他RNA富集純化
占比:Illumina RiboZero與Thermo RiboMinus系列使用該方法
優勢:磁珠法,操作簡單;可應用于混合樣本;
缺點:樣本起始量要求高(一般為1 μg);rRNA殘留量相對方法1略高;
流程圖:
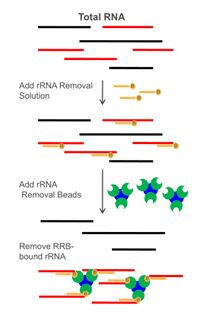
更多關于rRNA去除方法請點擊NGS mRNA建庫*攻略—從mRNA純化到注意事項
rRNA的“高效去除”
Hieff NGS® MaxUp rRNA Depletion Kit (Human/Mouse/Rat)利用RNase H消化法去除人、小鼠、大鼠總RNA中的核糖體RNA。該試劑盒對于完整和部分降解的總RNA(如FFPE RNA)均具有良好的rRNA去除效果。可用于高通量測序分析mRNA和非編碼RNA,也可用于cDNA合成或其它下游應用。
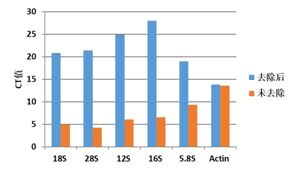
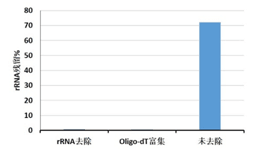
圖3:1μg 293T 總RNA 進行 rRNA 去除,利用 qPCR 對比去除前后 rRNA 基因和 mRNA 基因的Ct值變化(左圖)
分別以1μg 人、小鼠、大鼠總RNA為樣本,試劑盒去除rRNA后建庫,測序獲得rRNA殘留%數據(右圖)
產品信息:
| 產品名稱 | 貨號 | 規格 | *格(元) |
| Hieff NGS® MaxUp rRNA Depletion Kit (Human/Mouse/Rat) | 12253ES08 | 8T | 2530 |
| 12253ES24 | 24T | 9090 | |
| 12253ES96 | 96T | 29890 |
參考文獻:
- Adiconis X , Borges-Rivera D , Satija R , et al. Corrigendum: Comparative analysis of RNA sequencing methods for degraded or low-input samples[J]. Nature Methods, 2014, 11(2):210-210.
- Zhao W , He X , Hoadley K A , et al. Comparison of RNA-Seq by poly (A) capture, ribosomal RNA depletion, and DNA microarray for expression profiling[J]. Bmc Genomics, 2014, 15(1).
- Petrova O E , Garcia-Alcalde F , Zampaloni C , et al. Comparative evaluation of rRNA depletion procedures for the improved analysis of bacterial biofilm and mixed pathogen culture transcriptomes[J]. Scientific Reports, 2017, 7:41114.

 初級會員·13年
初級會員·13年
